

Nasze
"Małe Ojczyzny"
(miejscowości i regiony)
Rozmieszczenie nazwiska w Polsce w 2002 roku
Genetic genealogy of Oziembłowski (Oziębłowski) Families

Porównanie
autosomalnego DNA (at-DNA) 2 kuzynów
z rodu Oziembłowskich z Niżankowic w kontekście etniczno-geograficznym
osoby: "01at_Niżankowice" (zestaw 169070) oraz
"02at_Niżankowice" (zestaw 191848)
Comparision
of autosomal-DNA (at-DNA) of two cousins from Oziembłowski
Family in Niżankowice (Family Finder) in the ethnic-geographical context
Poniżej porównano wybrane fragmenty czterech chromosomów autosomalnych (nr 2, 3, 12, 15) dwóch osób urodzonych w Niżankowicach (Przemyśl) i będących w stosunku do siebie jednocześnie kuzynami w 4 i 6 pokoleniu co ukazano na schemacie na innej podstronie.
Wybór
określonych fragmentów czterech chromosomów miał miejsce w związku
z (połowicznym) "podobieństwem" tych regionów zarówno
u osoby "01at-Niżankowice" (zestaw 169070) jak i u osoby
"02at-Niżankowice" (zestaw 191848), co na poniższych rysunkach
(reprezentujących chromosomy nr 2, 3, 12 i 15) zaznaczone jest głównie
żółtymi paskami (nad paskami niebieskimi). Obszary chromosomów zaznaczone
na żółto oznaczają "połowiczną zgodność" par bazowych,
na zielono - "pełną zgodność", zaś na czerwono "brak
zgodności" pomiędzy adekwatnymi obszarami chromosomów u osoby
"01at-Niżankowice" oraz osoby "02at-Niżankowice".
Niebieskie paski oznaczają "podobne" segmenty chromosomów
(najczęściej połowicznie lub całkowicie) u obu osób, w przypadku
gdy "długość" tych segmentów jest większa niż 3 [cM]. |
 |
 |
 |
 |
 Na
jednej z podstron
GEDmatch.com można dokonać porównania wybranego chromosomu (1-22)
w odniesieniu do dwóch różnych stestowanych osób (Ad-Mix Utility). Zastosowany
tam algorytm jest mniej dokładny aniżeli DIYDodecad
V2 czy też DIYDodecad
V3, niemniej jednak wystarczający do ogólnego wnioskowania.
Na
jednej z podstron
GEDmatch.com można dokonać porównania wybranego chromosomu (1-22)
w odniesieniu do dwóch różnych stestowanych osób (Ad-Mix Utility). Zastosowany
tam algorytm jest mniej dokładny aniżeli DIYDodecad
V2 czy też DIYDodecad
V3, niemniej jednak wystarczający do ogólnego wnioskowania.
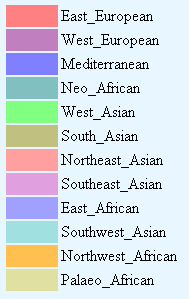
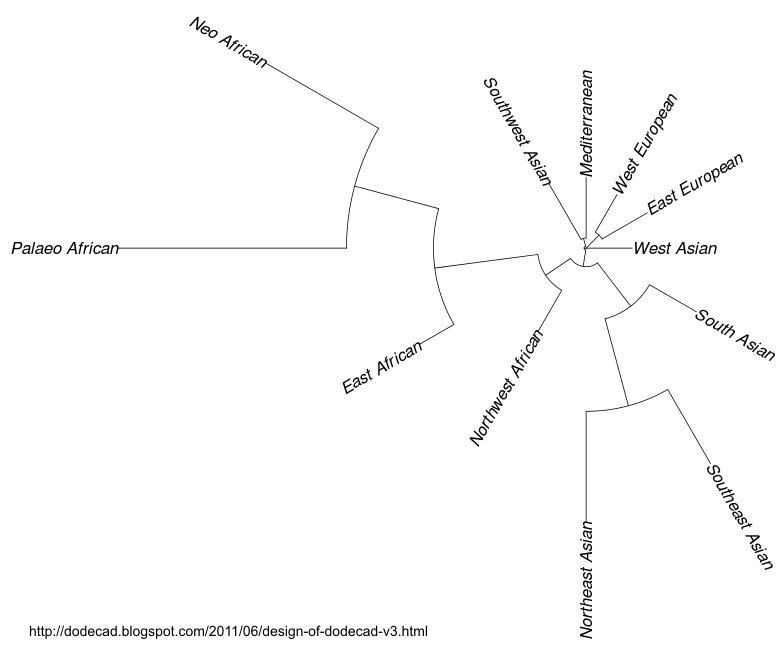
"Odniesieniem" jest 12 referencyjnych populacji nazwanych w sposób "geograficzny". Porównywane są "krok po kroku" wszystkie odpowiednie SNPy osoby stestowanej z utworzoną wcześniej bazą (na podstawie badań różnych populacji) i wykreślane są wąskie paski jedno, dwu lub więcej kolorowe (maksymalnie 12, gdyż jest 12 "populacji") jeden obok drugiego, dzięki czemu uzyskuje się wielokolorowy schemat danego chromosomu lub jego fragmentu. Konkretne kolory przypisane są do jednej z 12 referencyjnych populacji, tak jak zostało to zobrazowane na sąsiednim rysunku. Wzajemne zależności filogenetyczne referencyjnych populacji przedstawiono na poniższym schemacie pobranym ze strony opisującej DIYDodecad V3.
W pewnym uproszczeniu można powiedzieć, że populacja wschodnioeuropejska (East European) jest w podobnym stopniu podobna do populacji zachodnioeuropejskiej (West European) jak populacja śródziemnomorska (Mediterranean) do populacji Azji południowo-zachodniej (Southwest Asian). Z tego samego pnia "wyrasta" populacja zachodnioazjatycka (West Asian) tak więc ta piątka populacji jest genetycznie stosunkowo blisko siebie. Wyraźnie osobne "gałązki" tworzą 3 pozostałe populacje azjatyckie oraz cztery afrykańskie. Nie ma tutaj populacji natywnych mieszkańców Ameryki czy Australii. Większość zastosowanych określeń jest geograficznie jednoznaczna, niemniej jednak nazwy dwóch populacji afrykańskich nie są jednoznaczne i wymagają pewnego wyjaśnienia. Populacja "Neo African" odnosi się do populacji m.in. potomków przymusowych niewolników sprowadzanych do Ameryki głównie z Afryki Zachodniej (w tym Mandinka, rejon Senegalu). Określenie populacji "Paleo African" odnosi się zaś do mieszkańców Afryki Południowej (w tym do ludności "San" czy tzw. "Buszmenów").
12 populacji referencyjnych ukazanych na drzewku filogenetycznym
Poniżej przedstawiono odpowiednie (zazwyczaj "połowicznie zgodne") fragmenty czterech chromosomów kuzynów z Niżankowic. Na podstawie pobieżnego oglądu można stwierdzić, że na chromosomie nr 2 na najdłuższym zgodnym pomiędzy kuzynami segmencie o długości 35,8 [cM] dominują głównie fragmenty charakterystyczne dla populacji zachodnioeurpejskiej (West European) i w mniejszym stopniu dla populacji wschodnioeuropejskiej (East European).
Drugi pod względem długości segment (29,2 cM) występujący na chromosomie nr 15 nie posiada fragmentów jednoznacznie typowych dla jednej czy dwóch populacji, zwłaszcza że u jednego z kuzynów (osoba "01at-Niżankowice") występuje spory fragment charakterystyczny dla populacji śródziemnomorskiej, zaś u drugiego kuzyna w tym miejscu podobnej obserwacji nie poczyniono. Wspólne są za to są obserwacje dotyczące pozostałego fragmentu tego segmentu na 15 chromosomie gdzie występuje w miarę zrównoważony "mix" genetyczny charakterystyczny dla 3 populacji: wschodniosłowiańskiej, zachodniosłowiańskiej oraz zachodnioazjatyckiej.
Na chromosomie 15 jest jeszcze inny (nieco krótszy) "zgodny" pomiędzy kuzynami segment o długości 17,8 [cM], którego największe podobieństwo jest, jak się wydaje, do populacji śródziemnomorskiej.
Segment chromosomu 12 o łącznej długości 25,9 [cM] wydaje się być zgodny z populacją zachodnioeuropejską i mniej ze wschodnioeuropejską. U osoby "02at-Niżankowice" występuje fragment charakterystyczny dla Azji Południowej, którego to nie ma w tym miejscu u osoby "01at-Niżankowice".
Ostatni omawiany segment o długości 14,5 [cM] występujący na chromosomie nr 3 jest podobny u obu kuzynów i charakterystyczny dla populacji przede wszystkim zachodnioeuropejskiej oraz w mniejszym stopniu dla populacji zachodnioazjatyckiej i południowoazjatyckiej.
Wspólni przodkowie kuzynów z Niżankowic, jak się wydaje, mogli mieć sporo podobieństwa genetycznego do populacji zachodnioeuropejskiej i nieco mniej do populacji wschodnioeuropejskiej. Nie do pominięcia wydaje się fakt, że przodkowie ich mogli mieć relatywnie większe (niż stestowani potomkowie) podobieństwo do populacji azjatyckich, co byłoby konsekwencją niespokojnych czasów w dawnych wiekach, kiedy najazdy ze wschodu były zjawiskiem dość powszechnym.
Segment chromosomu nr 2 kuzynów z
Niżankowic: 35,8 [cM] i 5319 SNP-ów (wg GEDmatch.com) na odcinku ok. 220M - 240M (tj.: 220,730,216 - 240,353,275) |
Chr
2 Kit F191848  |
Chr 2 Kit
F169070 |
Segment
chromosomu nr 3 kuzynów z Niżankowic:
14,5 [cM] i 2013 SNP-ów (wg GEDmatch.com) na odcinku ok. 0M - 5M (tj.: 36,495 - 4,728,178) |
Chr 3
Kit F191848 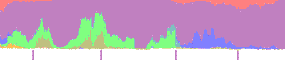 |
Chr 3
Kit F169070  |
Segment
chromosomu nr 12 kuzynów z Niżankowic:
17,1+8,8 [cM] i 3182+1873 SNP-ów (wg GEDmatch.com) na odcinku ok. 104M - 124M (tj.: 104,008,831 - 116,125,612 oraz 116,129,528 - 123,573,984) |
Chr 12
Kit F191848 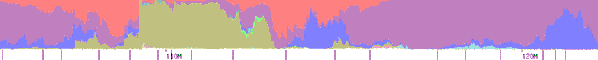 |
Chr 12
Kit F169070 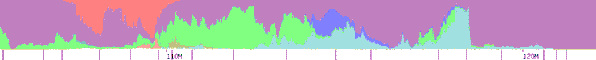 |
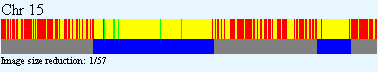
Segment
chromosomu nr 15 kuzynów z Niżankowic:
29,3 [cM] i 6817 SNP-ów (wg GEDmatch.com) na odcinku ok. 40M - 67M (tj.: 40,014,733 - 67,186,610) |
Chr 15
Kit F191848  |
Chr 15
Kit F169070  |
Segment
chromosomu nr 15 kuzynów z Niżankowic:
17,8 [cM] i 1889 SNP-ów (wg GEDmatch.com) na odcinku ok. 91M - 96M (tj.: 90,839,824 - 95,901,320) |
Chr 15
Kit F191848 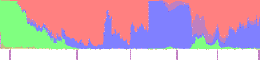 |
Chr 15 Kit F169070 |


