Nasze
"Małe Ojczyzny"
(miejscowości i regiony)
Rozmieszczenie nazwiska w Polsce w 2002 roku
Genetic genealogy Y-DNA of Oziembłowski (Oziębłowski) Families


I1-M253P (I1-ASP) z wykorzystaniem "skalowania wielowymiarowego" i "analizy skupień"
autor: Maciej Oziembłowski
Statistical investigation of STR Y-DNA haplotypes in I1-M253P (I1-ASP) subclade
using of "multidimensional scaling" and "cluster analysis"
Haplotypy dwóch Oziembłowskich (zestaw 169071 i 191847) są w praktyce identyczne (dla Y67) i zaliczone zostały do subkladu I1-M253P (I1-ASP). W dalszej części niniejszego tekstu występują one jako haplotyp "Oziembłowski" (I1-ASP). Więcej o samym subkladzie można znaleźć na mojej innej podstronie, gdzie jest on omówiony w ramach projektu FT-DNA "Normanowie Europy Kontynentalnej" (Normans-CE). Chciałem sprawdzić jak z wykorzystaniem standardowych technik statystycznych (pakiet Statistica 9.0) będą wyglądały "grupowania" haplotypów w ramach subkladu I1-ASP (według stanu na 10.10.2010) i skutkiem tego jest poniższy artykuł.
1) 20 haplotypów STR Y-DNA (67 markerowych) zostało pozyskanych z projektu Normans-CE zaś 2 haplotypy uzyskano z bazy danych Ysearch (Matz, Reck). Dwa ostatnie haplotypy były zakwalifikowane do tego subkladu przez Peter'a Gwozdz'a. Tak więc do badań wykorzystano w sumie 22 haplotypy. Wartości markerów STR Y-DNA dla tych 22 haplotypów (osób) znajdują się w Tab. 1 (trzy części: 1a, 1b, 1c).
2) W drugim etapie dokonano standaryzacji danych, aby markery o wysokich wartościach np. ok. 34-35 nie były w modelu "silniejsze" od tych markerów z niższymi wartościami, np. 8-9 (Tab. 2a i 2b). Ponieważ dla 22 markerów wartości były identyczne dla wszystkich haplotypów (osób), to ostatecznie po standaryzacji do dalszych badań wykorzystano wartości 45 markerów (67-22=45), dla których występowało jakiekolwiek zróżnicowanie w ramach tychże 22 haplotypów (osób).
3) Na podstawie Tab. 2 utworzona została macierz odległości (Tab. 3). Im większa występuje liczba dla danej pary haplotypów tym ma miejsce mniejsze ich podobieństwo genetyczne. Niższe liczby = większe podobieństwo genetyczne.
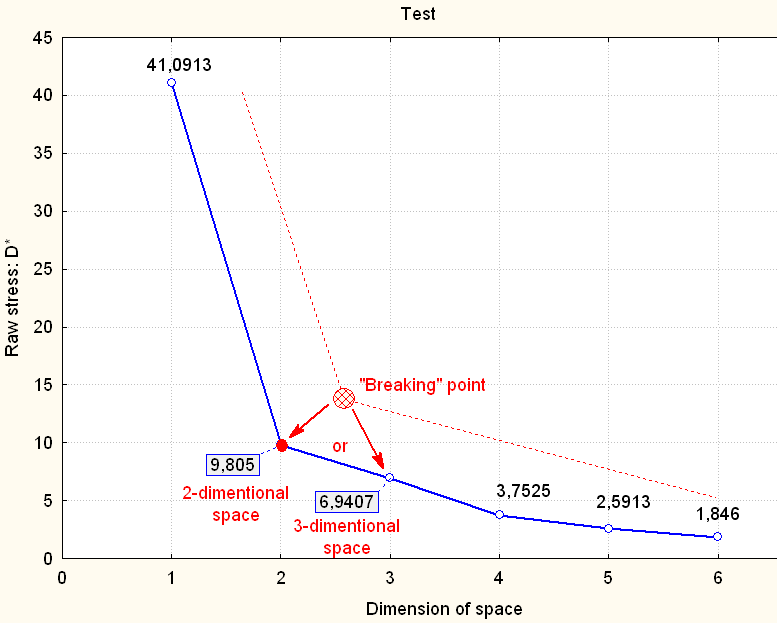
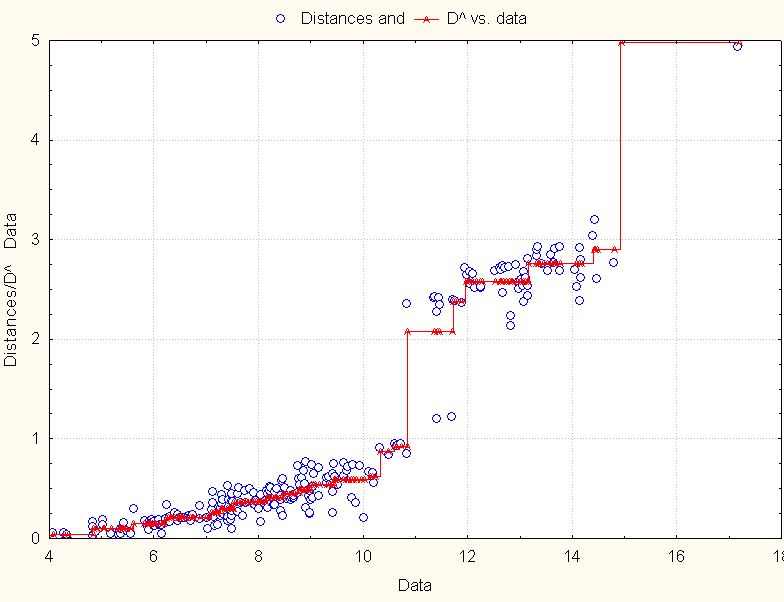
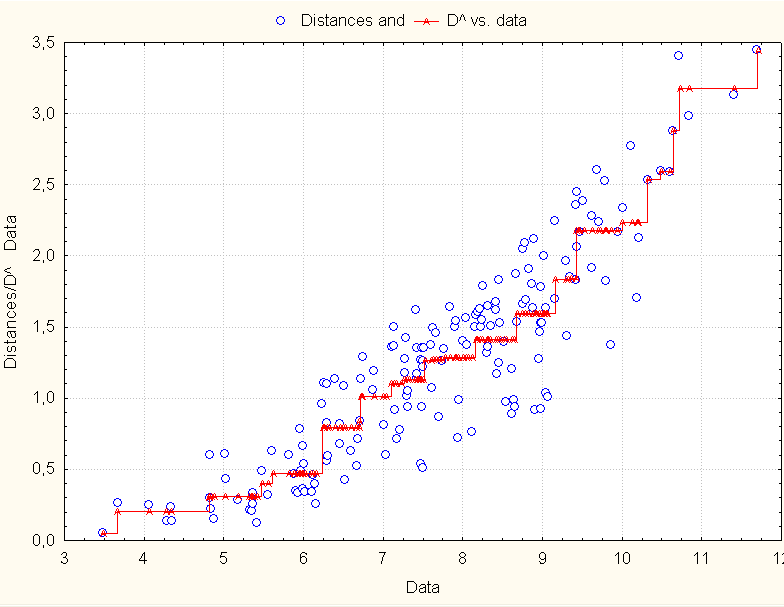
4) Macierz z Tab. 3 była wyjściową dla metody "Skalowanie wielowymiarowe". Wykres osypiska wskazuje, że model 2 lub 3-wymiarowy będzie dobrze opisywał zależności. Wybrano model 2-D (Rys. 1). Diagram Shepard'a (Rys. 2) potwierdza, że konfiguracja dwuwymiarowa w odpowiedni sposób opisuje podobieństwa pomiędzy analizowanymi haplotypami STR (punkty są blisko "schodków"). Każdy punkt reprezentuje jedną porównywaną parę haplotypów. W kilku przypadkach punkty "nachodzą" na siebie, ale wszystkich "par" dla 22 haplotypów jest 231 co wynika z dwumianu Newtona lub prościej - z obliczenia wyrażenia: (22*21)/2.
Rys.
1
Wykres osypiska (dla 22 osób=haplotypów)
Rys.
2
Diagram Shepard'a (dla 22 osób=haplotypów)
5) Model dwuwymiarowy, czyli płaszczyzna (Rys. 3) pokazuje wzajemne podobieństwa pomiędzy analizowanymi haplotypami. Im punkty reprezentujące dane haplotypy są bliżej siebie, tym większe jest ich podobieństwo genetyczne. Trzech uczestników ma mocno różniące się haplotypy od pozostałych 19 osób, dlatego punkty ich reprezentujące znajdują się w obszarze nazwanym przeze mnie "Far Outer Space".
Rys.
3
Podobieństwo 22 haplotypów dla modelu dwuwymiarowego 2D

6) "Przybliżając się" do pozostałych 19 punktów (zoom-in) widać, że można by dokonać manualnego podziału na dwie grupy (Rys. 4): "Middle Outer Space" oraz "Inner Space". Te 19 punkty są przesunięte nieco w prawo od początku układu współrzędnych (0,0), gdyż po lewej stronie znajdują się dwa punkty (Rys. 3) na "Far Outer Space", a po prawej tylko jeden, gdyż model uwzględnia wszystkie 22 punkty.
Rys.
4
Podobieństwo 22 haplotypów dla modelu dwuwymiarowego 2D
(ale "zoom-in" tylko na 19 haplotypów)

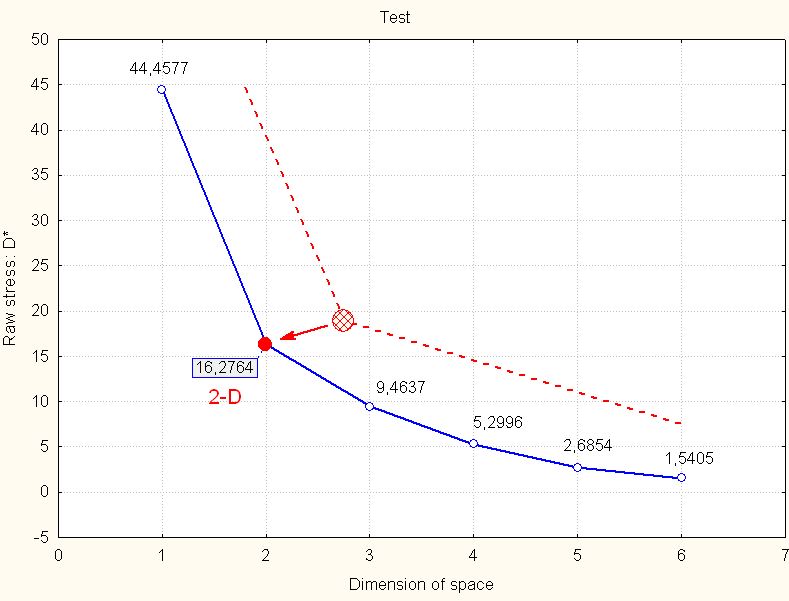
7) Dokonano wykluczenia z modelu 3 najbardziej "odstających" od reszty haplotypów. Dla pozostałych 19 powtórzono kroki: osypisko - model 2-D (Rys. 5), diagram Shepard'a (Rys. 6) - OK, bo punkty blisko schodków, par do porównania mniej: 171 = (19*18)/2 oraz na koniec wykreślono 2-wymiarowy model dla 19 profili (Rys. 7). Tutaj też utworzył się "rdzeń" (10 profili) w centralnym położeniu układu współrzędnych. W samym centrum jest profil osób: Bembnista, Pietruszewski. Najbliżej 10 osobowego rdzenia z "Middle outer space" jest Johnsen. Jest rzeczą oczywistą, że dodawanie lub odejmowanie z modelu określonych haplotypów wpływa na wzajemną konfigurację "punktów" na płaszczyźnie, gdyż każdy haplotyp coś do ogólnego modelu "wnosi" lub "zabiera" jeśli go wykluczamy.
Rys.
5
Wykres osypiska (dla 19 osób=haplotypów)
Rys.
6
Diagram Shepard'a (dla 19 osób=haplotypów)
Rys.
7
Podobieństwo 19 haplotypów dla modelu dwuwymiarowego 2D

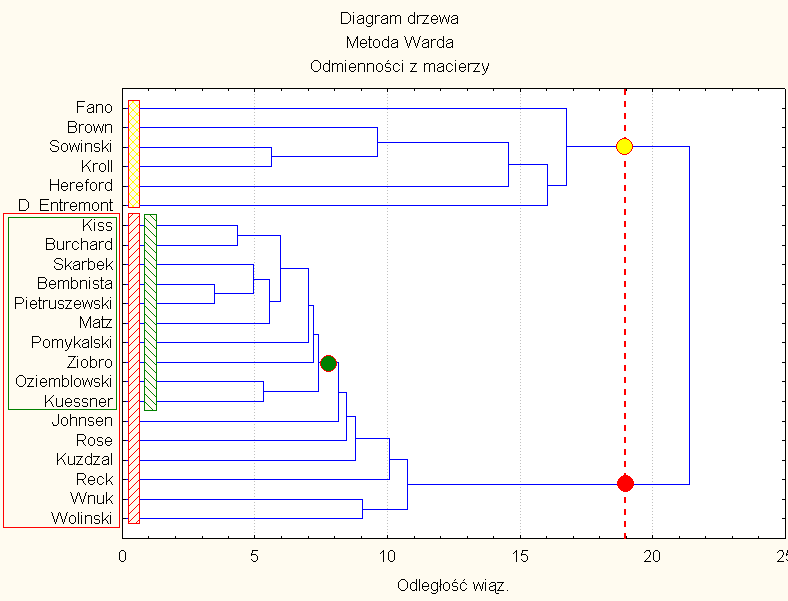
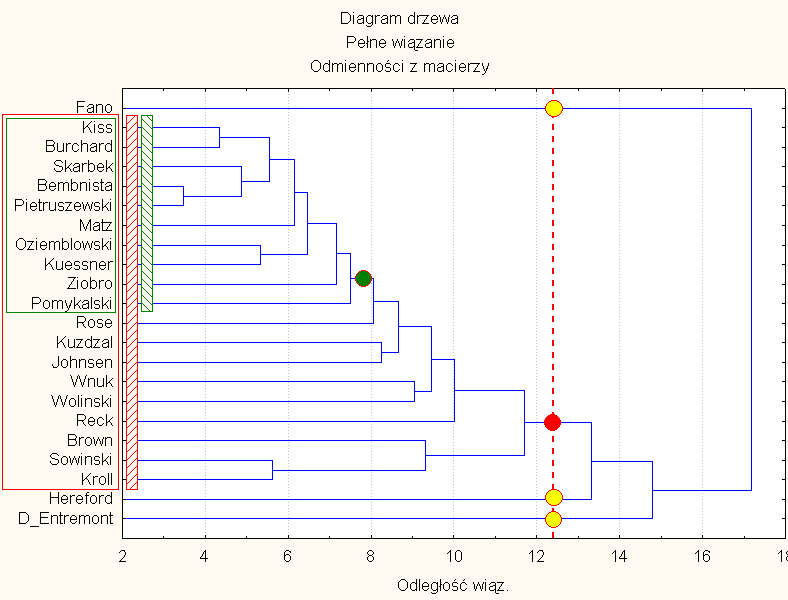
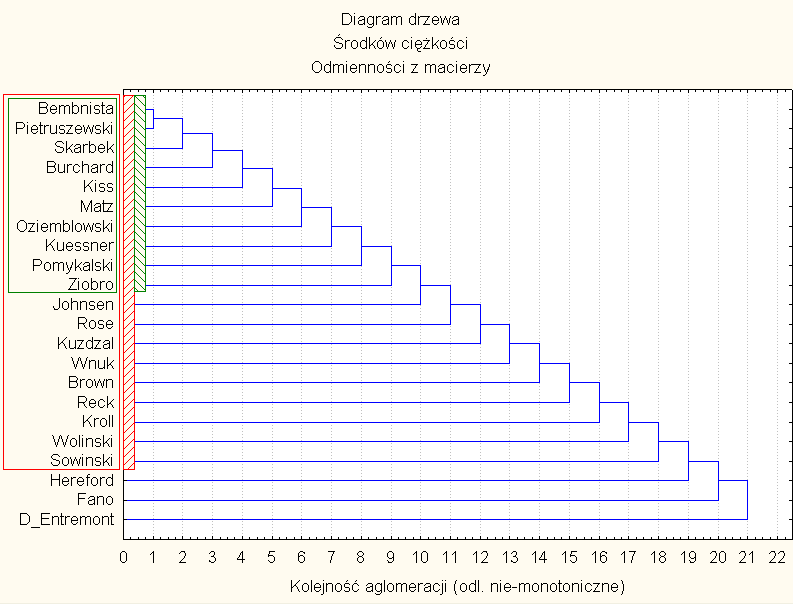
9) Wykreślono 5 diagramów drzewa (Rys. 8-12) gdzie zastosowano nieco różniące się metody "wiązania", a mianowicie metodę wiązania pojedynczego (Rys. 8), wiązania pełnego (Rys. 9), średnich połączeń (Rys. 10), środków ciężkości (Rys. 11) oraz metodę Warda (Rys. 12). Diagramy zasadniczo potwierdzają obserwacje z metody skalowania wielowymiarowego, ale należy pamiętać, że w obu metodach materiałem wyjściowym była ta sama macierz.
Rys.
8
Podobieństwo 22 haplotypów w metodzie "wiązania pojedynczego"

Rys.
9
Podobieństwo 22 haplotypów w metodzie "wiązania pełnego"
Rys.
10
Podobieństwo 22 haplotypów w metodzie "średnich połączeń"

Rys.
11
Podobieństwo 22 haplotypów w metodzie "środków ciężkości"
Rys.
12
Podobieństwo 22 haplotypów w metodzie "Ward'a"