Nasze
"Małe Ojczyzny"
(miejscowości i regiony)
Rozmieszczenie nazwiska w Polsce w 2002 roku
Genetic genealogy Y-DNA of Oziembłowski (Oziębłowski) Families


I1-M253P (I1-ASP) na podstawie "analizy filogenetycznej"
autor: Maciej Oziembłowski
Haplotypy
dwóch Oziembłowskich (zestaw 169071 i 191847)
będąc w praktyce identyczne (dla Y67) zaliczone zostały do subkladu
I1-M253P (I1-ASP). W dalszej części na rysunkach występują one jako
haplotyp z nazwą "Oziembłowski" (I1-ASP). Więcej o samym subkladzie
można znaleźć na mojej innej podstronie, gdzie jest
on omówiony w ramach projektu FT-DNA "Normanowie Europy Kontynentalnej"
(Normans-CE).
Celem niniejszej pracy było sprawdzenie jak z wykorzystaniem "analizy
filogenetycznej" będą wyglądały "grupowania" 22 haplotypów
Y67 w ramach subkladu I1-ASP (Projekt
FT-DNA "Normans CE" według stanu na 22.04.2011). Poszczególne
kroki niniejszej analizy oparte zostały na metodzie
opisanej przy projekcie genetycznym Y-DNA nazwiska Ham (autorstwa
Dave'a Hamm'a), która to procedura opisana jest również na
Histmag.org.
1) 22 haplotypy STR Y-DNA (67 markerowych) zostały pozyskane z projektu FT-DNA Normans-CE. Wartości markerów STR Y-DNA dla tych 22 haplotypów (osób) znajdują się w Tab. 1 (trzy części: 1a, 1b, 1c).
2) W drugim etapie dokonano analizy oraz konwersji danych z Tab. 1 na format Phylip. Wykorzystano do tego program online, zaś dane w formacie Phylip zapisano jako plik txt.
3) Dane w formacie Phylip (punkt 2) poddane zostały dalszej analizie za pomocą programu "Kitsch" (dostępnego darmowo w internecie). W porównaniu do domyślnych ustawień programu włączono funkcję "Lower Triangular data matrix?" z "no" na "yes" (klawisz "L") oraz klawiszem "J" zmieniono funkcję "Randomize input order of species?" na następujące wartości: "Random number seed": 9 oraz "Number of times to jumble": 99 [podane wartości ze względu na czas analizy są sensowne gdy analizuje się np. dwadzieścia kilka haplotypów, większa liczba haplotypów = mniejsza druga wartość]. Efektem pracy programu są dwa pliki: "outfile" i "outtree". Można je otworzyć np. windowsowym notatnikiem i zapisać w formacie txt.
Plik
txt outfile
Plik
txt outtree
Drzewo filogenetyczne subkladu I1-ASP (w prostej postaci) znajduje się w pliku outfile, niemniej jednak plik outtree może służyć innym programom jako plik z danymi.
4) Plikowi outtree (z punktu 4) można ręcznie dopisać rozszerzenie "tre". Plik taki będzie rozpoznawany przez darmowy program MEGA5, dzięki któremu wykreślić można różne drzewa filogenetyczne (ukazujące to samo, ale w różny graficznie sposób). Poniżej ukazano tylko wybrane typy drzew filogenetycznych spośród wielu możliwych do wykreślenia z wykorzystaniem wspomnianego programu.
Rys.
1
Podobieństwo 22 haplotypów ustalone na podstawie analizy filogenetycznej
(drzewo filogenetyczne - domyślne)

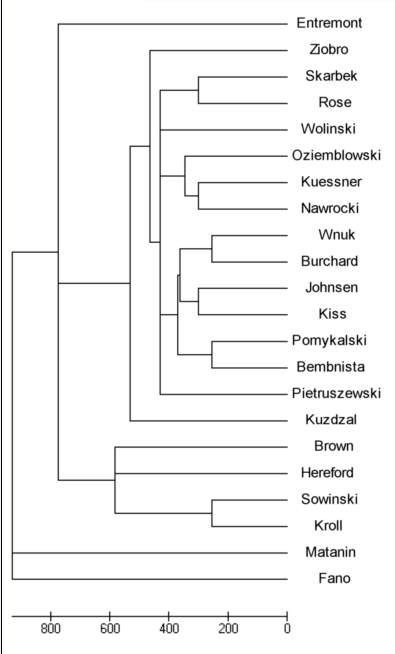
Rys.
2
Podobieństwo 22 haplotypów ustalone na podstawie analizy filogenetycznej
(drzewo filogenetyczne - Compute Linearized Tree)
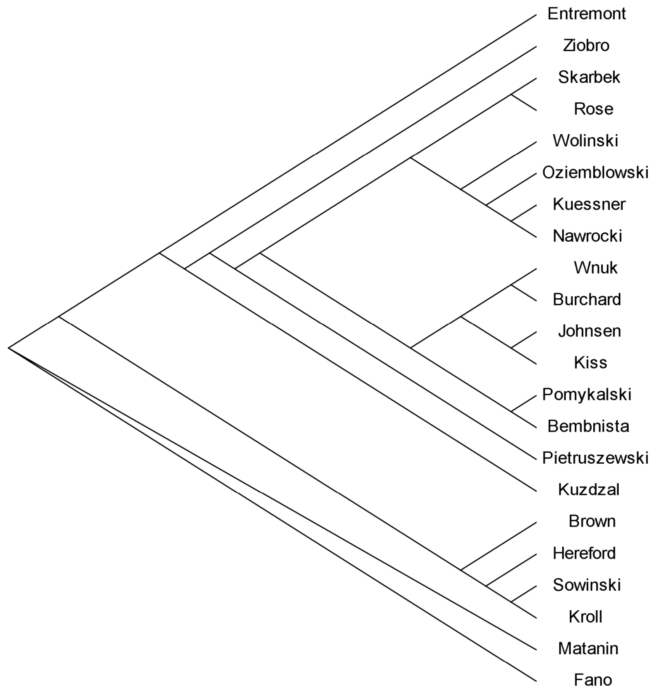
Rys.
3
Podobieństwo 22 haplotypów ustalone na podstawie analizy filogenetycznej
(drzewo filogenetyczne - Tree/Branch Style > traditional > straight
[topology only])
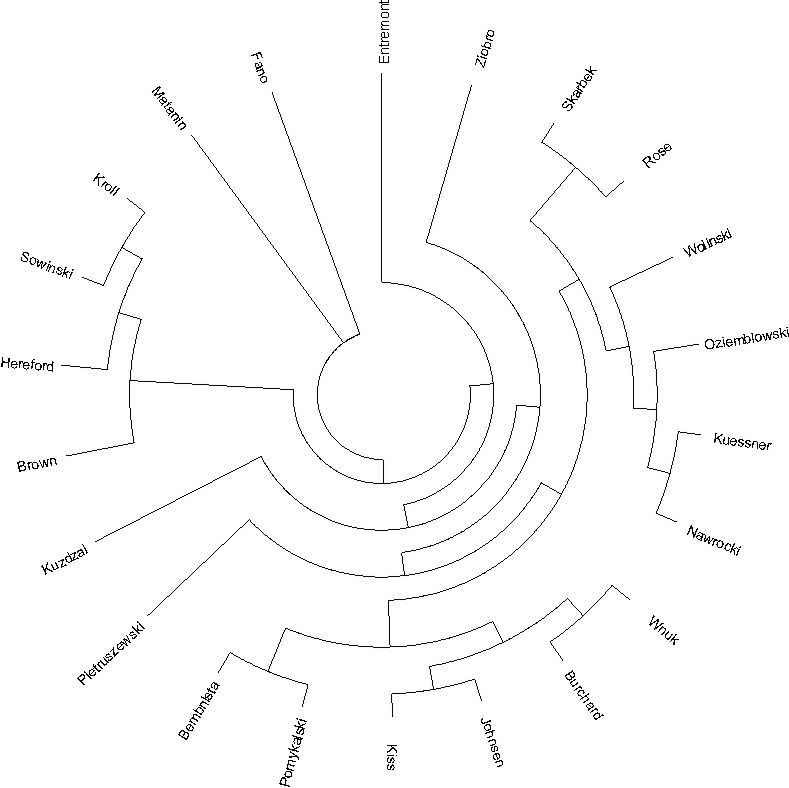
Rys.
4
Podobieństwo 22 haplotypów ustalone na podstawie analizy filogenetycznej
(drzewo filogenetyczne - Tree/Branch Style > circle [topology only])